classify
Clasificar observaciones mediante análisis discriminante
Sintaxis
Descripción
Nota
Se recomienda utilizar fitcdiscr y predict en lugar de classify para entrenar un clasificador de análisis discriminante y predecir etiquetas. fitcdiscr admite la validación cruzada y la optimización de hiperparámetros, y no requiere que ajuste el clasificador cada vez que haga una nueva predicción o cambie las probabilidades a priori.
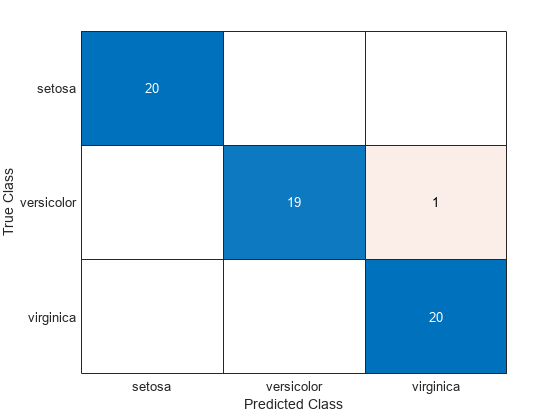
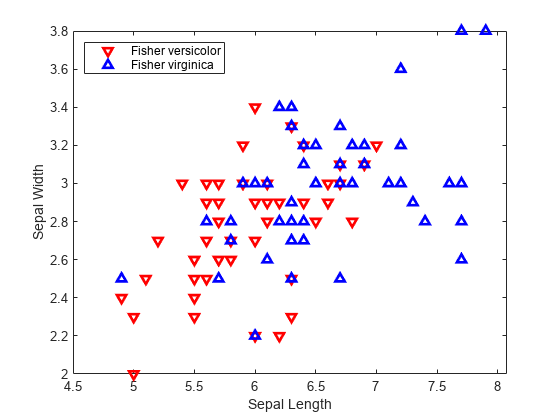
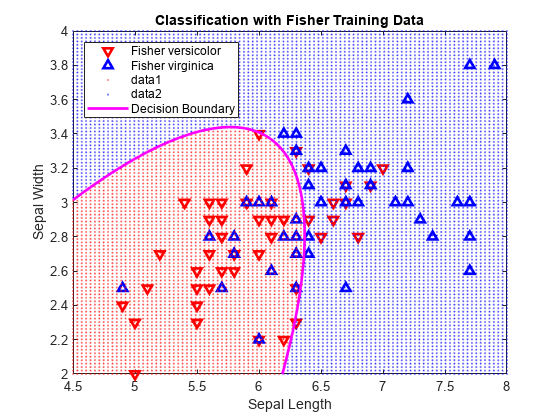
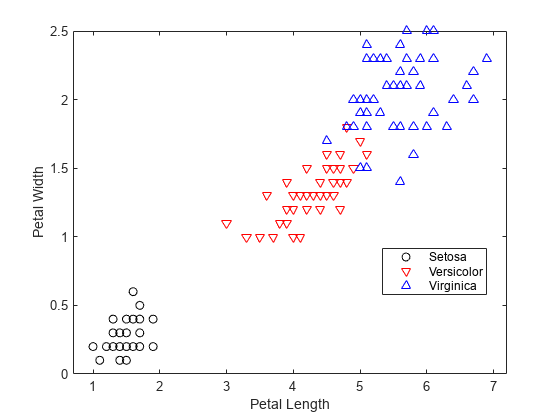
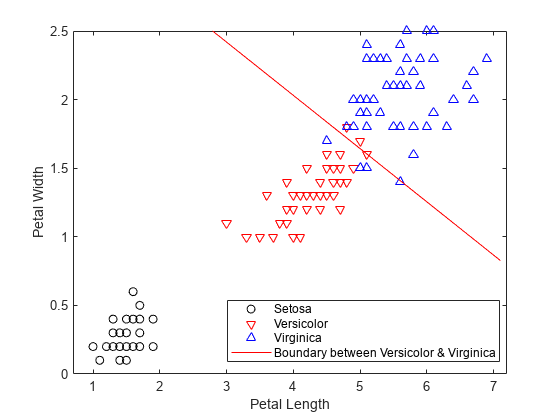
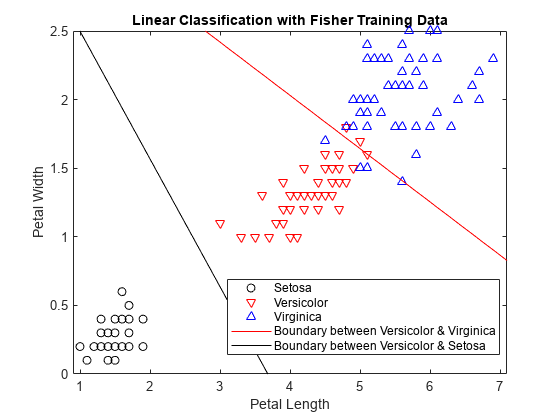
class = classify(sample,training,group)sample en uno de los grupos a los que pertenecen los datos de training. Los grupos de training se especifican mediante group. La función devuelve class, que contiene los grupos asignados para cada fila de sample.
[ también devuelve la tasa de error aparente (class,err,posterior,logp,coeff] = classify(___)err), las probabilidades a posteriori de las observaciones de entrenamiento (posterior), el logaritmo de la densidad de probabilidad incondicional de las observaciones de muestra (logp) y los coeficientes de las curvas de límite (coeff), utilizando cualquiera de las combinaciones de argumentos de entrada de las sintaxis anteriores.
Ejemplos
Argumentos de entrada
Argumentos de salida
Funcionalidad alternativa
La función fitcdiscr también realiza un análisis discriminante. Puede entrenar un clasificador con la función fitcdiscr y predecir las etiquetas de los nuevos datos con la función predict. La función fitcdiscr admite la validación cruzada y la optimización de hiperparámetros, y no requiere que ajuste el clasificador cada vez que haga una nueva predicción o cambie las probabilidades a priori.
Referencias
[1] Krzanowski, Wojtek. J. Principles of Multivariate Analysis: A User's Perspective. NY: Oxford University Press, 1988.
[2] Seber, George A. F. Multivariate Observations. NJ: John Wiley & Sons, Inc., 1984.
Historial de versiones
Introducido antes de R2006a